LIME for ECG Time Series Dataset Example
LIME (Local Interpretable Model-Agnostic Explanations) — популярный модет в решении задачи интерпретации. Он основан на простой идее — приблизить прогнозы сложного оценщика (например, нейронной сети) простым — обычно линейной/логистической регрессией.
Применить LIME можно из коробки при помощи одноименной библиотеки [lime [1]](https://github.com/marcotcr/lime [1]). Однако, при применении LIME к, в частности, к временным рядам возникают особенности:
-
При интерпретации нужно учесть, что временные ряды — это структурированные последовательности.
-
Интерпретация проводится не по отдельным признакам, а по сегментам времени: насколько важен отрезок сигнала с 20-й по 30-ю миллисекунду?
-
Для генерации соседних примеров нужно аккуратно вносить изменения в части сигнала, соответствующие сегментам: заменой на шум, среднее значение или другие методы.
поэтому в чистом виде lime для TimeSeries не всегда легко применить. И в этом туториале мы сделаем приближенение метода самостоятельно! :)
А именно, вы:
-
Узнаете, как использовать LIME для интерпретации моделей классификации сигналов ЭКГ;
-
Разберете способ генерации осмысленных локальных возмущений;
-
Обучите локальную модель, имитирующую поведение [2] основной нейросети;
-
Визуализируете, какие сегменты ЭКГ сигнала оказались наиболее значимыми для классификации.
Работать будем с PyTorch + Scikit-learn. Весь код, как всегда, будет на гитхаб.
Step 0. Loading and Preprocessing the Dataset
Работать будем с набором данных про ЭКГ. А именно, в нашем распряжении будут записи ЭКГ по четырем классам:
-
Нормальный синусовый ритм;
-
Мерцательная аритмия;
-
Желудочковая тахикардия;
-
Сердечный приступ.
Более подробная информация про датасет [здесь [3]].
# Path to the dataset
file_path_train = 'https://github.com/SadSabrina/XAI-open_materials/blob/main/LIME_for_Time_Series/data/ecg_train.csv?raw=True'
file_path_test = 'https://github.com/SadSabrina/XAI-open_materials/blob/main/LIME_for_Time_Series/data/ecg_test.csv?raw=True'
# Training dataset
ecg_train = pd.read_csv(file_path_train, header=None)
# Testing dataset
ecg_test = pd.read_csv(file_path_test, header=None)Сделаем несколько шагов классической предобработки — посмотрим на пропуски, бесконечности и уберем их. Затем, поделим данные на X и y, подберем метрику и приступим к подготовке модели. Все эти шаги описаны в ноутбуке, на них подробно останавливаться не будем.
Step 1. Building a CNN Model
Дальше, оформим модель, на основе которой будем решать задачу. На самом деле, в силу данных, можно было бы обойтись интерпретируемой моделью. Но так как цель туториала — практика в интерпретации черных ящиков — решим задачу при помощи сверточной сети.
class ECGCNN(nn.Module):
def __init__(self, input_shape, num_classes):
"""
input_shape: tuple time_steps, channels (exmaple 140, 1)
num_classes: number of output classes
"""
super(ECGCNN, self).__init__()
time_steps, channels = input_shape
self.conv1 = nn.Conv1d(in_channels=channels, out_channels=64, kernel_size=3)
self.pool = nn.MaxPool1d(kernel_size=2)
self.dropout = nn.Dropout(0.5)
# Compute output length after conv and pooling
conv_output_length = time_steps - 2 # kernel_size=3 reduces by 2
pool_output_length = conv_output_length // 2 # MaxPool1d(pool_size=2)
self.flattened_size = 64 * pool_output_length
self.fc1 = nn.Linear(self.flattened_size, 100)
self.fc2 = nn.Linear(100, num_classes)
def forward(self, x):
"""
x: Tensor of shape (batch_size, time_steps, channels)
"""
if type(x) != torch.Tensor:
x = torch.Tensor(x)
x = x.permute(0, 2, 1) # Change to (batch_size, channels, time_steps)
x = F.relu(self.conv1(x))
x = self.pool(x)
x = self.dropout(x)
x = x.contiguous().view(x.size(0), -1) # Flatten
x = F.relu(self.fc1(x))
x = self.fc2(x)
return torch.log_softmax(x, dim=1) # Use CrossEntropyLoss, so no softmax here (pyTorch CrossEntropyLoss expects unbounded scores)
def forward_probs(self, x):
x = self.forward(x)
return torch.exp(x)
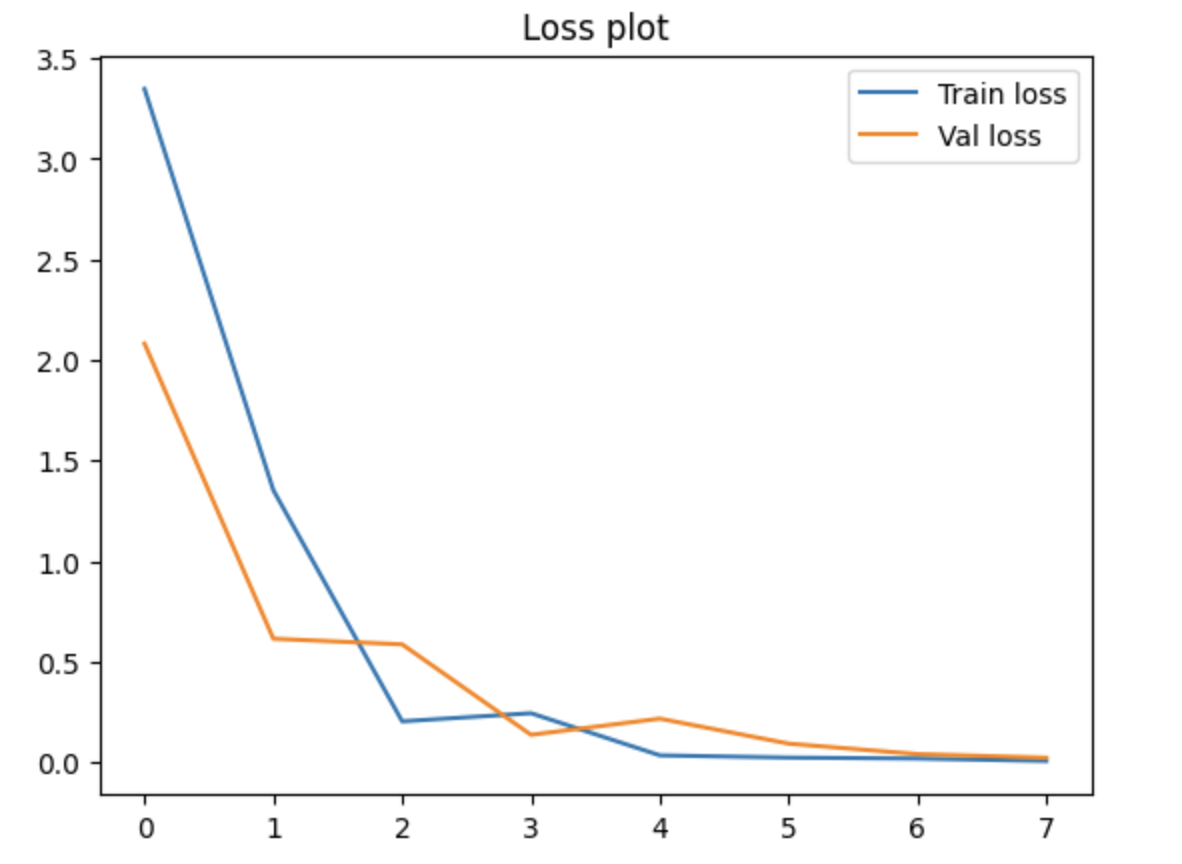
Обучение [4] даст нам такой график, так что теперь приступим к LIME!
Step 2. LIME for Time Seris Domain
По определению, объяснение LIME — это локальная интерпретируемая модель, которая аппроксимирует поведение [5] исходной сложной модели в окрестности конкретного объекта $x in mathbb{R}^d$ ($d$ — количество признаков в объекте).
Математически [6] это задача оптимизации:
где:
-
— сложная (исходная) модель, например нейросеть;
-
— интерпретируемая модель (обычно линейная), G красивая здесь — это множество всех интепретируемых моделей;
-
— вес, отражающий близость объекта
к
; обычно вычисляется как:
, где D — расстояние (например, косинусное),
— ширина ядра;
-
— локальная функция потерь, измеряющая насколько хорошо
приближает
в окрестности
— мы будем брать MSE;
-
— мера сложности интерпретируемой модели (например, число ненулевых коэффициентов).
На практике, чтобы использовать эту формулу, нам нужны:
-
Конкретное наблюдение
– его будем объяснять;
-
То, что мы будем считать за признаки во временном ряду (в нашем случае семгенты временного ряда);
-
Окрестность — множество наблюдений
вокруг
;
-
Прогнозы —
на вариантах из полученной окресности;
-
Обученная интерпретируемая модель
на точках из окресности (обученная с учётом весов
), веса которой и будут отражать важность;
Соберем всё по шагам.
Шаг 1. Конкретное наблюдение
Здесь можно взять любой индекс и забрать его из датасета. У нас будет 11й.
idx_ecg = 11 # сhoose interested index from rows
instance_ecg = ecg_test_x.iloc[idx_ecg, :].values # extract the selected instance from the test dataset
plt.figure(figsize=(16, 5))
plt.plot(instance_ecg)
plt.title('ECG instance to explain');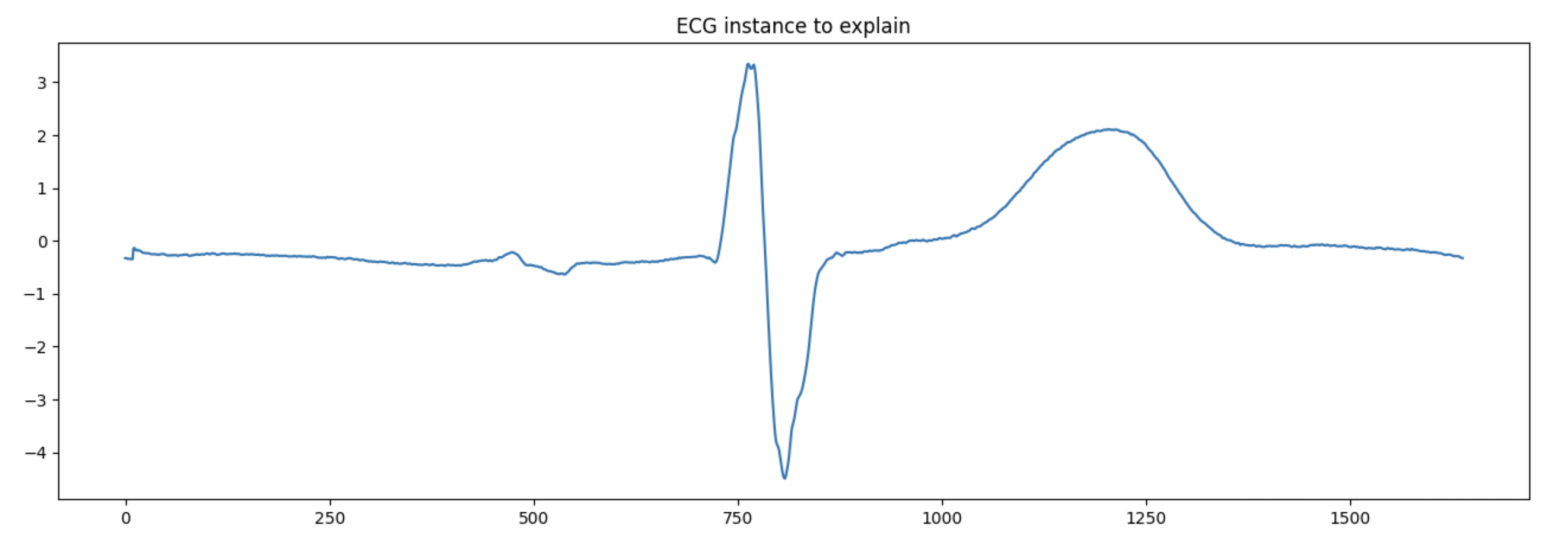
Получим для него вероятности и класс.
# Predict the class of the selected signal by trained model
probability_vector = model.forward_probs(torch.Tensor(np.expand_dims(instance_ecg.reshape(1, -1), axis=2)))
print("Probability vector of the selected instance:", probability_vector)
# Probability vector of the selected instance: tensor([[3.3614e-06, 8.0830e-01, 1.9169e-01, 1.8729e-06]],
# grad_fn=<ExpBackward0>)
# class labels
class_labels = [0, 1, 2, 3]
predicted_class = torch.argmax(probability_vector).item()
print("Available classes:", class_labels)
print("Predicted Class for the selected instance:", predicted_class)
# Available classes: [0, 1, 2, 3]
# Predicted Class for the selected instance: 1Шаг 2. Получение сегментов-признаков.
Методом “пристального взгляда” подберем количество сегментов. Более правильный и формальный способ — подбирать количество сегментов в зависимости от природы сигнала.
Сегменты получим простым образом — нарезкой ряда на равные кусочки. А именно, для ряда с временными шагами
мы делим его на
отрезков фиксированной длины:
где — длина одного сегмента.
Дальше мы строим индексы границ отрезков следующим образом:
Где последний сегмент может включать “хвост”:
Это нужно, чтобы даже если длина ряда не делится на
нацело, последний сегмент корректно включит все оставшиеся точки.
L = len(instance_ecg) # TS length
K = 13 # number of slices
l = L // K # slice width
# Segment start points
segment_edges = [i * l for i in range(K)] + [L]
# Segment centers
segment_centers = [(segment_edges[i] + segment_edges[i+1]) // 2 for i in range(K)]
# Segment labels
segment_labels = [f'{i+1}' for i in range(K)]
# Plot segmented instance
plt.figure(figsize=(12, 3))
plt.plot(instance_ecg, label='Original signal')
for i in range(1, K):
plt.axvline(x=i*l, color='r', linestyle='--')
plt.xticks(ticks=segment_centers, labels=segment_labels, fontsize=9)
plt.title('Segmented the instance ECG signal')
plt.xlabel('Segment index')
plt.ylabel('Signal Amplitude')
plt.legend()
plt.show()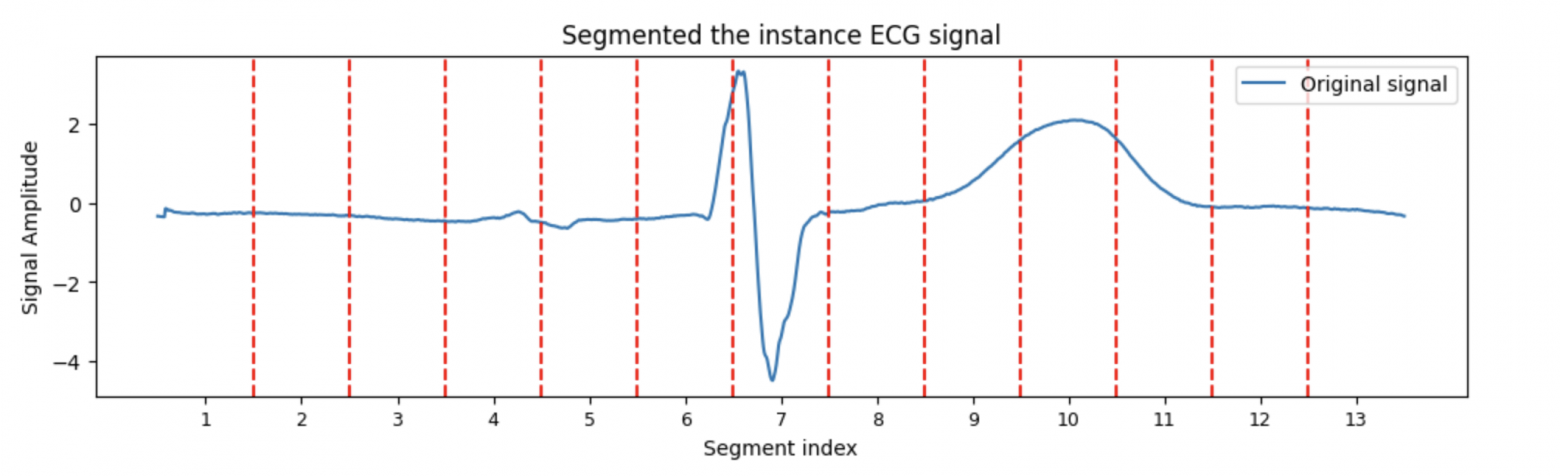
Шаг 3. Генерация наблюдений в окресности.
Будем генерировать наблюдения из окресности следующим образом. Для изначального наблюдения будем выбирать случайные номера сегментов
, и вносить в них изменения по одной из трех стратегий:
-
“mean” — меняем значения сегмента на среднее по сегменту;
-
“noise” — меняем значения сегмента на значения из нормального распределения, со средним и стандартным отклонением по сегменту;
-
“zero” — просто обнуляем значения сегмента
Все замены будем проводить по кусочкам .
# Segment slices function
def split_series(series, num_slices):
length = series.shape[0]
slice_len = int(np.ceil(length / num_slices))
return [(i * slice_len, min((i + 1) * slice_len, length)) for i in range(num_slices)]
slices = split_series(instance_ecg, 13)
def generate_perturbations(series, slices, num_samples, replacement="mean", pertub_power=0.2):
perturbed = []
masks = []
for _ in range(num_samples):
mask = np.ones(len(slices), dtype=int)
idx_to_perub = np.random.choice(len(slices), size=int(len(slices)*pertub_power), replace=False) # менять за раз будем pertub_power% от всех кусочков
mask[idx_to_perub] = 0
copy = series.copy()
for i in idx_to_perub:
start, end = slices[i]
if replacement == "mean":
copy[start:end] = np.mean(series[start:end])
elif replacement == "zero":
copy[start:end] = 0
elif replacement == "noise":
copy[start:end] = np.random.normal(series[start:end].mean(), series[start:end].std(), end - start) #.reshape(1, -1)
perturbed.append(copy)
masks.append(mask)
return np.array(perturbed), np.array(masks)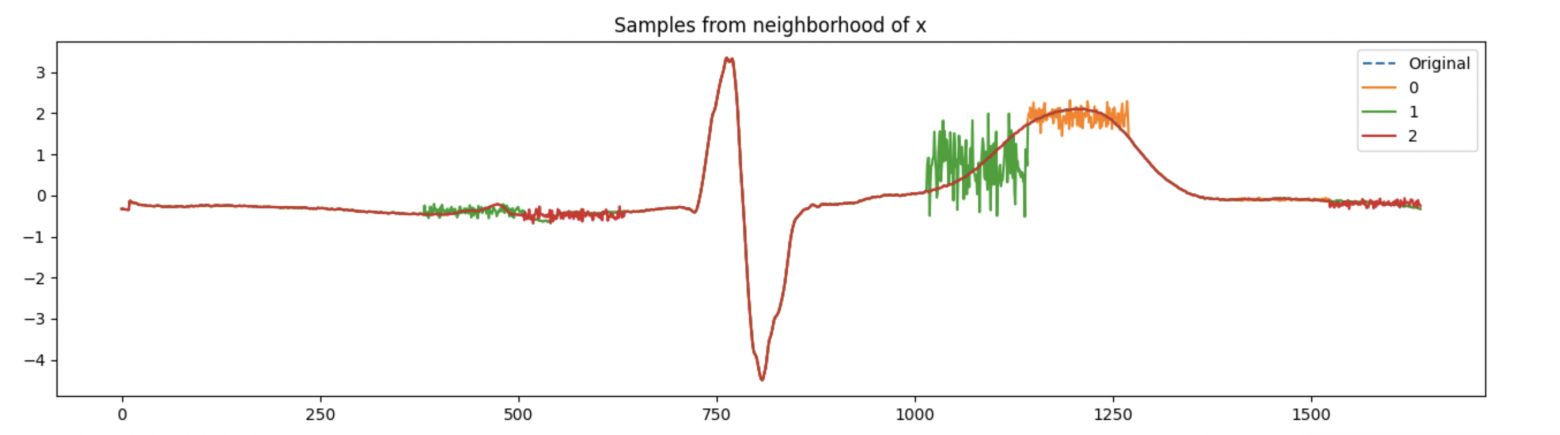
Обратите внимание [7], на этом шаге мы получими маски (masks). Для обучения интерпретируемой модели набор данных будет представлен бинарными признаками для каждого сегмента. Признак равен 1, если соответствующий сегмент включен, и 0, если он был искажен. Такой подход используется также при реализации LIME для [текстовых данных [8]].
Шаг 4. Обучение интерпретируемой модели в окресности.
Чтобы обучить модель будем, вместо решения исходной задачи, решать задачу обеспечения похожести интерпретируемой модели на модель-черный ящик. Для этого будем обучать модель давать прогнозы такие же, какими их дает исходная модель.
Обучать модель будем на вероятностях и в соответствие с важностью наблюдения. А именно, чем ближе он к исходному объекту, тем весомее он должен быть при обучении.

# Probabilities for all observations from the neighborhood
probs = np.exp(model(np.expand_dims(pertub, axis=2)).detach().numpy())
# Calculate the distances
distances = cosine_distances(pertub, instance_ecg.reshape(1, -1)).ravel()
def train_lime_ridge(perturbations, predictions, distances, target_class, alpha=1.0, kernel_width=0.25):
"""
Train interpretable model (Ridge)
Parameters:
perturbations (np.array): features [N x K], mask
predictions (np.array): матрица [N x C], вероятности классов
distances (np.array): длина N, расстояния до оригинала
target_class (int): индекс класса, который объясняется
alpha (float): коэффициент регуляризации Ridge
kernel_width (float): ширина ядра для весов
Returns:
(weights, intercept, score): коэф. модели, свободный член, R^2 на подмножестве
"""
# LIME exponential kernel
weights = np.exp(- (distances ** 2) / (kernel_width ** 2))
# target
y = predictions[:, target_class]
# simple Ridge-regression train
model = Ridge(alpha=alpha)
model.fit(perturbations, y, sample_weight=weights)
return model.coef_, model.intercept_, model.score(perturbations, y, sample_weight=weights)
w, b, score = train_lime_ridge(masks, probs, distances, 0, alpha=.8)B теперь, когда у нас есть модель, мы можем использовать её веса для интерпретации основной модели по сегментам. Сделаем это в виде двух графиков. Код для отрисовки приложен в ноутбуке.
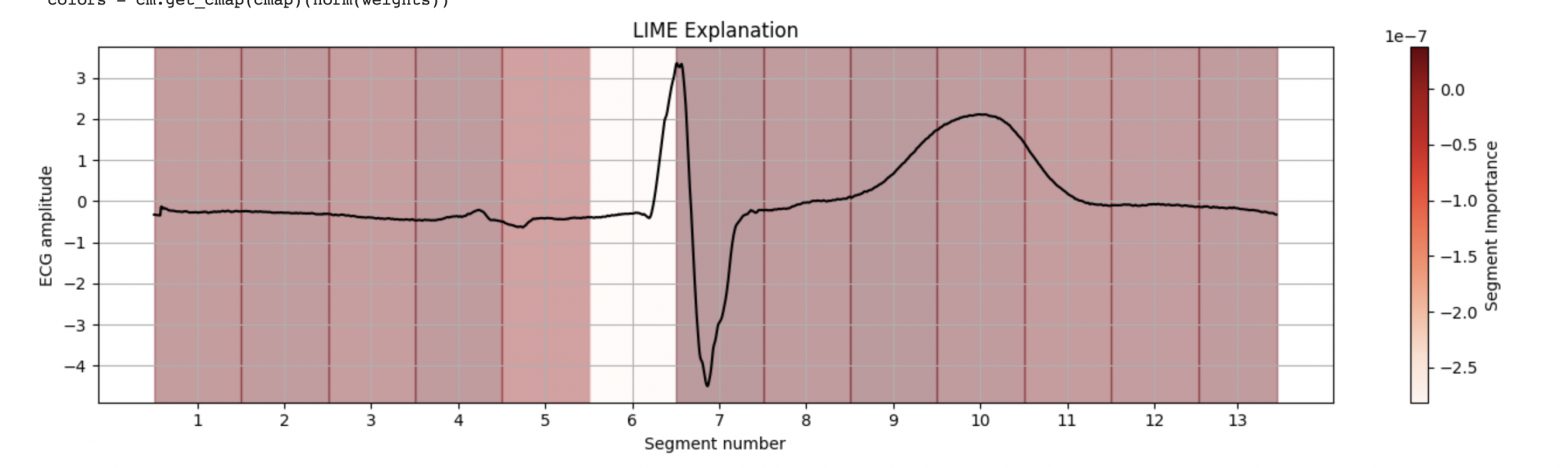
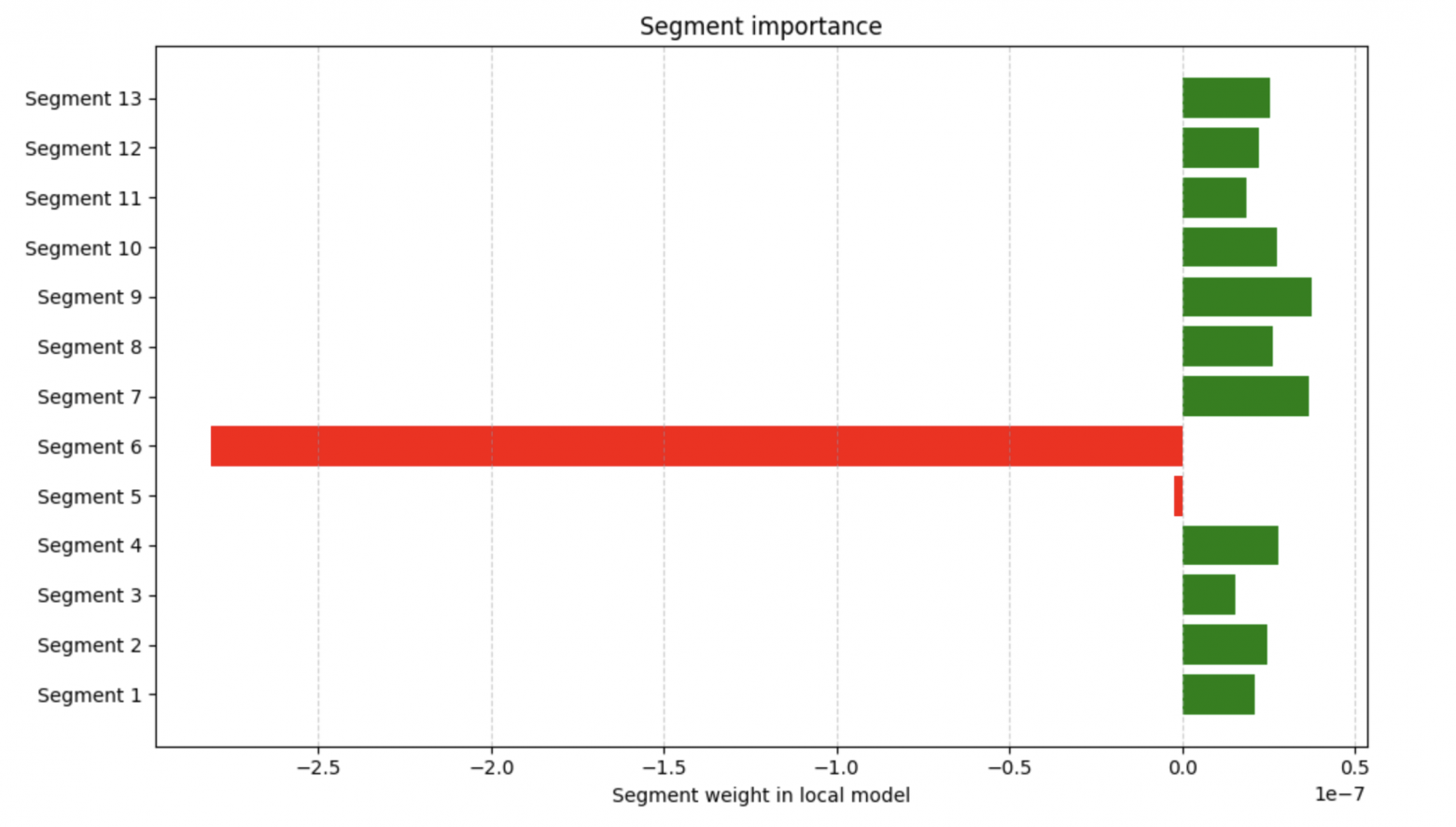
Готово! Спасибо, друзья!
В этом туториале мы рассмотрели применение метода LIME к задаче интерпретации модели классификации временных рядов на примере ЭКГ-сигналов. Для этого:
-
обучили простую сверточную нейросеть для распознавания классов ЭКГ;
-
реализовали механизм генерации локальных объяснений с помощью сегментации временного ряда;
-
построили визуализации важности сегментов как поверх сигнала, так и в виде диаграммы весов.
LIME позволил нам локально аппроксимировать поведение модели и оценить, какие части сигнала оказали наибольшее влияние на её предсказание.
Несмотря на простоту реализации, LIME остаётся мощным и гибким инструментом для анализа моделей с любыми типами входных данных. Однако стоит помнить, что качество интерпретации зависит от выбора параметров (например, числа сегментов) и стратегии генерации “окрестности” (можете поиграть также с параметрами функций и оценить насколько пошатнутся веса).
Основные материалы туториала:
[1](https://github.com/mdhabibi/LIME-for-Time-Series/tree/main?tab=readme-ov-file [9])
[2](https://github.com/emanuel-metzenthin/Lime-For-Time/tree/master [10])
[3](https://arxiv.org/abs/1602.04938 [11]).
Новые туториалы:
За ними всегда жду вас в [дата-блоге [12]]!
Обещаю публиковать материалы чаще! :)
Со всем самым добрым,
Ваш Дата-автор!
Автор: sad__sabrina
Источник [13]
Сайт-источник BrainTools: https://www.braintools.ru
Путь до страницы источника: https://www.braintools.ru/article/17083
URLs in this post:
[1] lime: https://github.com/marcotcr/lime
[2] поведение: http://www.braintools.ru/article/9372
[3] здесь: https://iopscience.iop.org/article/10.1088/1361-6579/abc960
[4] Обучение: http://www.braintools.ru/article/5125
[5] поведение: http://www.braintools.ru/article/5593
[6] Математически: http://www.braintools.ru/article/7620
[7] внимание: http://www.braintools.ru/article/7595
[8] текстовых данных: https://christophm.github.io/interpretable-ml-book/lime.html?utm_source=chatgpt.com
[9] https://github.com/mdhabibi/LIME-for-Time-Series/tree/main?tab=readme-ov-file: https://github.com/mdhabibi/LIME-for-Time-Series/tree/main?tab=readme-ov-file
[10] https://github.com/emanuel-metzenthin/Lime-For-Time/tree/master: https://github.com/emanuel-metzenthin/Lime-For-Time/tree/master
[11] https://arxiv.org/abs/1602.04938: https://arxiv.org/abs/1602.04938
[12] дата-блоге: https://t.me/jdata_blog
[13] Источник: https://habr.com/ru/articles/926082/?utm_campaign=926082&utm_source=habrahabr&utm_medium=rss
Нажмите здесь для печати.
