Разделяй и властвуй: отличие классов генеративных и предсказательных моделей в биоинженерии и NLP
De Novo дизайн молекул против предсказания текста: fight!
Возможно, факты покажутся очевидными, но я сознательно проговариваю их отдельно, потому что в сети смешиваются разные классы моделей — уверена, что и вы постоянно сталкиваетесь с утверждением, мол, «ИИ не способен создать ничего нового, модели только перерабатывают существующие данные».
Это мнение особенно распространено в контексте больших языковых моделей (LLM), GPT-подобных систем.
Но.
Биоинженерия сейчас входит в топовые тренды 2024-2025 как часть технологического «суперцикла» ИИ.
И применять мнение, упомянутое выше, к области молекулярного дизайна, неверно. Картина полностью меняется.
Что если я вам покажу, что генеративные модели действительно создают новые молекулы и структуры, ранее не существовавшие?
Сначала кратенько и грубо, а ниже разверну:

Пайплайн «как это часто собирают на практике»:
Это типичный модульный пайплайн. Компоненты могут существовать отдельно, естественно.
Генератор кандидатов => (опционально) фильтры/скоринг предикторами => отбор => лабораторная/физическая проверка.
Интерес [1] к этой теме возник в рамках моего проекта (я изучаю нейродегенеративные и генетические заболевания). Актуальные направления ИИ в сфере биоинженерии включают молекулярный дизайн, анализ белков, РНК [2] и разработку терапевтических молекул для новых лекарств.*
Пометка на полях: *я обобщила. По сути — генерация новых кандидатов (белки/биомолекулы) и их проверка экспериментально.
И когда в дискуссиях звучит заезженный тезис о том, что ИИ — это всего лишь «поиск по данным» или инструмент без творческого потенциала, критически важно уточнить контекст.
Тема очень актуальна в 2025 году.
ИИ в молекулярном дизайне и «drug discovery» — чуть ли не самое быстрорастущее направление: после AlphaFold 3 (2024) и новых генеративных моделей типа Evo (2024–2025), RFdiffusion All-Atom, Chroma и тд.
Выходит куча публикаций в Nature, Science и Nature Medicine. Плюс фармкомпании активно внедряют это (Insilico Medicine, Exscientia, Generate:Biomedicines и тд.). Общественный интерес «может ли ИИ изобретать новое» в связи с этим тоже растёт, особенно на фоне споров про креативность LLM.
В этой статье я попробую пояснить, почему ИИ в биодизайне способен генерировать инновационные структуры, и проведу небольшое сравнение с известными текстовыми моделями.
Часто под «ИИ» подразумевают исключительно LLM, игнорируя другие классы моделей, решающие принципиально иные задачи.
В биоинженерии/молекулярном дизайне различия между моделями особенно заметны
Давайте рассмотрим два основных типа моделей в этой области: предсказательные и генеративные (а потом вернёмся к LLM).
Предсказательные модели — анализируют существующие молекулы и прогнозируют их свойства.
В подмножество могут входить:
-
регрессоры (стабильность, аффинность);
-
классификаторы (токсично/нет);
-
дискриминативные модели в строгом смысле (которые учатся отличать одно от другого, например, больной белок от здорового; моделируют p(y|x))
Ещё уточнение во избежание недопониманий: в этом тексте я называю предсказательными любые модели, которые по заданной молекуле/последовательности оценивают свойства или класс; на практике это часто дискриминативные модели (классификаторы/регрессоры).
Они ничего не создают —берут существующую молекулу и говорят нам, как она себя поведет:
-
Как свернётся белок по данной аминокислотной последовательности? (AlphaFold от Google DeepMind – ИИ для предсказания структур белков (protein folding) и шире — применение ИИ в биологии)
-
Будет ли это токсично?
-
Свяжется ли лекарство с целью (аффинность, имею ввиду)?
То есть объект уже существует (хотя бы как последовательность), и мы про него узнаем что-то новое. Потому что релевантных взаимосвязей — космическое количество, человек просто не в состоянии все их увидеть и обработать вручную.
И есть генеративные модели
Вот они-то как раз работают в режиме De Novo Design — создают совершенно новые последовательности, которых раньше не существовало: генерация аминокислотных цепей, молекул, структур (соединений).
И ещё одно примечание. Что я имею ввиду под «не существовало»: последовательности и разные конструкции, которые не встречались в известных наборах природных последовательностей и проектируются De Novo под заданные ограничения и функции, а затем проходят экспериментальную проверку, я ещё раз это проговариваю.
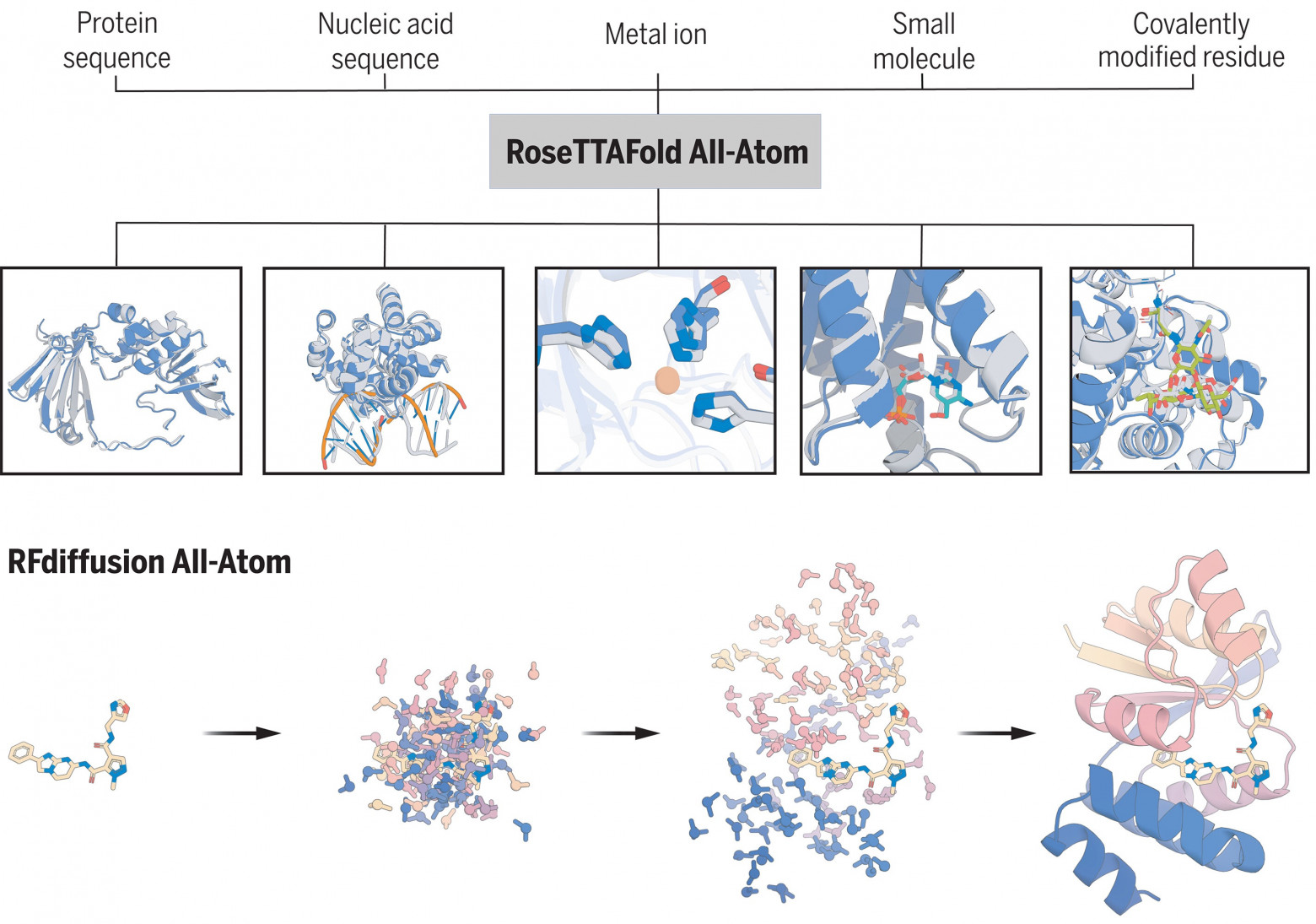
Тут я упомяну RFdiffusion (RoseTTAFold Diffusion) и его варианты, обязательно ProGen, Chroma и другие диффузионные модели (существует уже много подобных лабораторий).
Сгенерированные структуры проверяются экспериментально в лабораториях с участием людей, конечно же. Многие недавние публикации в Nature и Science основаны на таких подходах. Это активное направление исследований.
И здесь я выведу ключевой аспект различия!
Объективные законы физики и химии — структура либо стабильна и функциональна, либо нет
Это отличает процесс от чисто статистической генерации обычных NLP: если LLM галлюцинирует, то мы получаем бредовый текст, но если био-модель «галлюцинирует» (нарушает законы физики), то белок просто не свернется или распадется.
Ремарка:
в биодизайне «ошибка генерации» быстро проявляется: дизайн не проходит физико-химические фильтры или лабораторную валидацию, то есть невалидная/нефункциональная конструкция, провал в эксперименте, риск токсичности и тому подобное. И обычно у биоинформатиков терминология такого рода: invalid / non-physical / non-functional / failed design.
В биоинженерии есть «Hard Reality Check», которого нет у копирайтинга (успех De Novo дизайна экспериментально подтверждается рентгеноструктурным анализом или крио-ЭМ, например).
Лиричное уточнение:
Я не пытаюсь принизить LLM. Показываю, что ИИ бывает разным. LLM — это роскошный максимум: талантливый гуманитарий, а AlphaFold/RFdiffusion — это физик-инженер.
Вернемся к нашим ̶б̶а̶р̶а̶н̶а̶м̶ LLM по типу GPT-подобных моделей. В них деление на предсказательные и генеративные менее выражено.
Архитектурно LLM решают задачу предсказания следующего токена. На уровне поведения это приводит к генерации текста. Предсказание и генерация здесь — две стороны одной медали.
Это всегда генеративная авторегрессивная модель, но генеративность достигается через предсказание токена.
Модели опираются на статистические закономерности в данных обучения/весах. Они комбинируют элементы неожиданным образом, но не выходят за пределы своего многообразия и не открывают новые фундаментальные законы (пока), и если говорить не упрощая, то не имеют встроенного механизма проверки гипотез реальностью и без внешних инструментов/экспериментов обычно воспроизводят/перекомбинируют статистические закономерности текста.
Да, у современных LLM внушительные надстройки над трансформером: RAG, дополнительное внимание [3], расширенное контекстное окно, динамика в контексте, RLHF, цепочки рассуждений. Плюс эмбеддинговое пространство постоянно растёт. Модель может комбинировать известные ей вещи в непредвиденных сочетаниях (какая-то эмерджентность минимальная), подхватывая и развивая идеи пользователей, но при всём этом она не может синтезировать то, чего в её пространстве принципиально не было; она генерирует внутри распределения, заданного обучением [4] и контекстом, и «новизна» там – комбинаторная, не физически-экспериментальная, как у генеративок в биодизайне.
Существуют и гибриды — модели, сочетающие черты обоих подходов
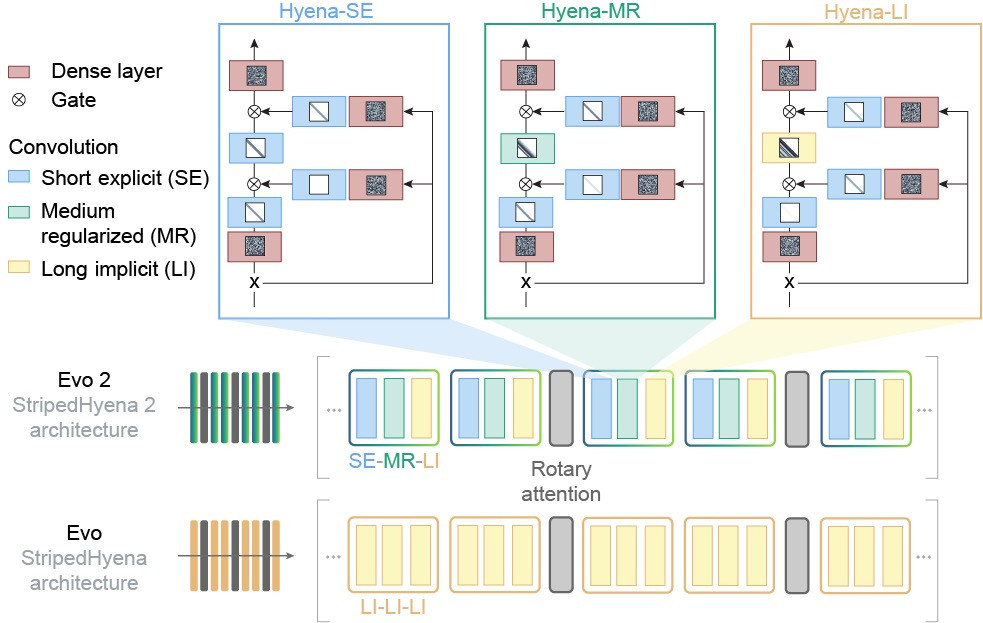
Пример — Evo (и Evo 2, 2024–2025 годов), геномная языковая модель (GLM), обученная на длинных последовательностях ДНК, РНК и белков в гигантских масштабах. Она предсказывает мутации и генерирует новые геномные последовательности, способна проектировать новые биологические системы, включая лабораторную проверку синтетических систем CRISPR (вероятно, вы слышали о «биологических» ножницах для ДНК). Для Evo «языком» служат как раз последовательности ДНК/РНК/белков в противовес обычным NLP, где фигурирует «человеческий» текст.
В Evo, по сути, тоже вкладывают биологические промпты (если позволите такой термин) — условия/ограничения и представление объекта (последовательность, мотивы, скелет, контактные карты…), которыми задаём пространство поиска.
Evo моделирует биологические ограничения (физика, химия, эволюция [5]), чтобы вывод был правдоподобным.
Разница с моделями NLP в нескольких аспектах: домене данных (текст vs биологические последовательности/структуры), целевой функции/валидации (человеческая правдоподобность vs физико-химическая/экспериментальная проверка), типе ограничений и тестов.
Те, кто говорит, что «ИИ ничего нового не создаёт» скорее всего смешивает класс моделей. В обработке текста генерация ограничена статистикой данных (веса, вероятности), в молекулярном дизайне — создаёт новые структуры, подчинённые законам природы.
Разделение этих подходов позволяет точнее оценивать возможности ИИ. В биоинженерии уже есть практические результаты: новые кандидаты в лекарства и синтетические белки.
Направление, считаю, перспективное. Оно продолжает развиваться.
Моя статья не претендует на полный обзор архитектур моделей. Целью ставила развести термины «генерация» и «предсказание» в разных доменах, потому что их часто путают, говоря об ИИ в целом.
Ещё мне бы хотелось вам показать статьи, заслуживающие внимания, чтобы не быть голословной. И несколько картинок.
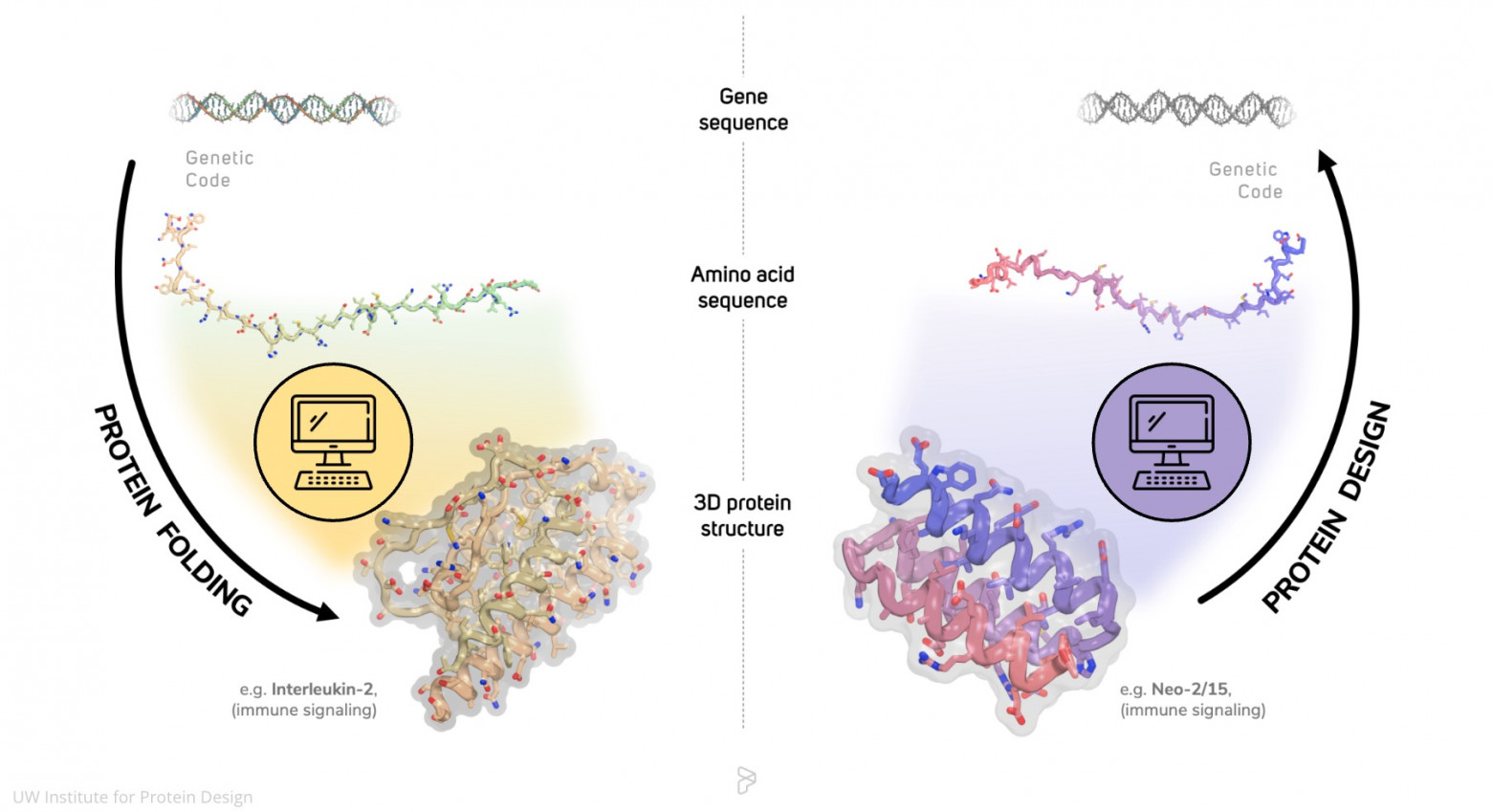
Для RFdiffusion (самый яркий пример De Novo с кучей публикаций в Nature): De Novo design of protein structure and function with RFdiffusion [7] (Nature, 2023) — тут генерация новых белков с нуля, включая функциональные (биндеры, энзимы и т.д.), с лабораторной валидацией.
Atomically accurate de novo design of antibodies with RFdiffusion [8] (Nature, 2025) — эта свежая, от пятого ноября: De Novo дизайн антител на атомном уровне.
Ещё продемонстрирую в качестве иллюстрации одной из работ вот такую последовательность

ProGen (показывают генерацию функциональных белков De Novo): Large language models generate functional protein sequences across diverse families [10] (Nature Biotechnology, 2023).
Здесь напрямую демонстрируют, что ProGen создаёт искусственные белки с предсказуемой функцией, и проверяют их экспериментально.
И ещё ProGen2: Exploring the boundaries of protein language models [11] (Cell Systems, 2023) —
(это, конечно, не Nature/Science, но тоже солидный журнал)
Про упомянутую мной выше Evo (который геномный De Novo)
Sequence modeling and design from molecular to genome scale with Evo [12] (Science, 2024) — генерация и дизайн на уровне геномов.
И Semantic design of functional De Novo genes from a genomic foundation model [13] (Nature, 2025) — Прямо про function-guided De Novo дизайн генов с Evo.
Illuminating protein space with a programmable generative model (Nature, 2023) — вот тут про Chroma [15], ещё одну генеративную модель для De Novo белков и комплексов.
[16] Не менее интересная модель, кстати.
И прикреплю ещё обзор LLM в биологии и химии [17]
Благодарю за прочтение!
Автор: II_shnitsa
Источник [18]
Сайт-источник BrainTools: https://www.braintools.ru
Путь до страницы источника: https://www.braintools.ru/article/23215
URLs in this post:
[1] Интерес: http://www.braintools.ru/article/4220
[2] РНК: http://www.braintools.ru/article/8114
[3] внимание: http://www.braintools.ru/article/7595
[4] обучением: http://www.braintools.ru/article/5125
[5] эволюция: http://www.braintools.ru/article/7702
[6] bakerlab.org: https://www.bakerlab.org/2023/10/30/introducing-all-atom-versions-of-rosettafold-and-rfdiffusion/
[7] De Novo design of protein structure and function with RFdiffusion: https://www.nature.com/articles/s41586-023-06415-8
[8] Atomically accurate de novo design of antibodies with RFdiffusion: https://www.nature.com/articles/s41586-025-09721-5
[9] science.org: https://science.org/doi/10.1126/science.adl2528
[10] Large language models generate functional protein sequences across diverse families: https://www.nature.com/articles/s41587-022-01618-2
[11] И ещё ProGen2: Exploring the boundaries of protein language models: https://www.sciencedirect.com/science/article/pii/S2405471223002727
[12] Sequence modeling and design from molecular to genome scale with Evo: https://www.science.org/doi/10.1126/science.ado9336
[13] Semantic design of functional De Novo genes from a genomic foundation model: https://www.nature.com/articles/s41586-025-09749-7
[14] developer.nvidia.com: https://developer.nvidia.com/blog/understanding-the-language-of-lifes-biomolecules-across-evolution-at-a-new-scale-with-evo-2/
[15] вот тут про Chroma: https://www.nature.com/articles/s41586-023-06728-8
[16]
: https://www.nature.com/articles/s41586-023-06728-8%EF%BF%BC
[17] И прикреплю ещё обзор LLM в биологии и химии: https://arxiv.org/abs/2401.14656
[18] Источник: https://habr.com/ru/articles/977042/?utm_campaign=977042&utm_source=habrahabr&utm_medium=rss
Нажмите здесь для печати.