-
Часть II: Скоринг, off-target и работа с реальными данными (вы тута)
В первой части мы разобрались с базовой биологией CRISPR-Cas9 и написали CLI-утилиту для поиска gRNA. Фильтровали по GC-составу и наличию повторов. В итоге получился рабочий, но очень простой инструмент.
Сегодня добавим функций: научим программу сортировать кандидатов по качеству и проверять, не порежет ли наша gRNA что-нибудь лишнее в геноме. А чтобы было интереснее прогоним всё на настоящем гене с криминальной историей.
Почему CCR5?
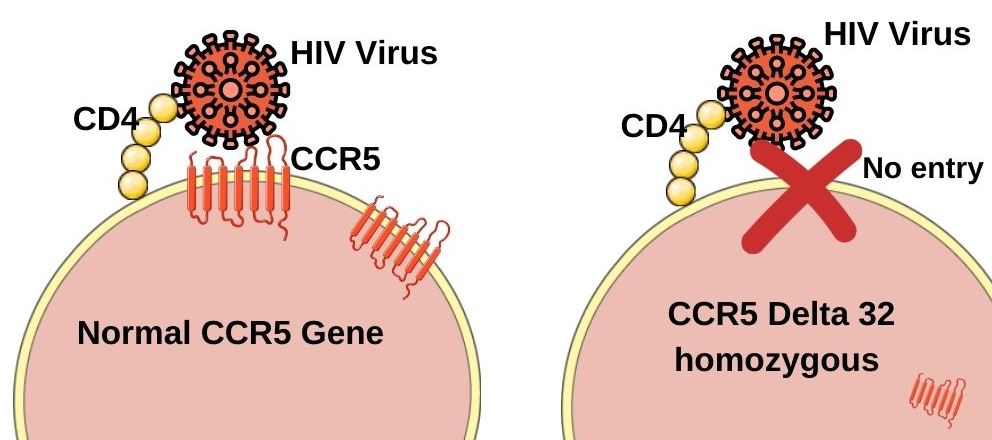
Помните скандал 2018 года? Китайский учёный Хэ Цзянькуй отредактировал геном человеческих эмбрионов и довёл беременность до родов. На свет появились близнецы Лулу и Нана – первые генетически модифицированные люди. Учёный получил три года тюрьмы, а научное сообщество – этическую мигрень на десятилетие вперёд.
Хэ целился в ген CCR5. Этот ген кодирует белок-рецептор на поверхности иммунных клеток – это что-то вроде дверной ручки, за которую ВИЧ хватается, чтобы проникнуть внутрь. Примерно у 1% европейцев есть мутация CCR5-Δ32: у них эта “ручка” сломана и, как следствие, такие люди устойчивы к ВИЧ от рождения.
Идея Хэ была в том, чтобы искусственно “сломать” CCR5 у эмбрионов и сделать детей невосприимчивыми к вирусу. Этика эксперимента – это отдельный разговор, но ген CCR5 с тех пор стал, пожалуй, самой известной мишенью для CRISPR.
Его и возьмём для тестов.
Ликбез: 5′ и 3′ – что за штрихи?
Прежде чем лезть в код, разберёмся с одной штукой, которая постоянно мелькает в биоинформатике.
ДНК – это цепочка нуклеотидов (A, C, T, G), связанных через сахарофосфатный “скелет”. Каждый сахар в этом скелете – молекула дезоксирибозы с пронумерованными атомами углерода: 1′, 2′, 3′, 4′, 5′ (читается “один штрих”, “пять штрих”). Штрих – чтобы не путать с номерами атомов в азотистых основаниях.
Цепь ДНК всегда имеет направление, как улица с односторонним движением:
-
5′-конец – начало цепи, там торчит свободная фосфатная группа
-
3′-конец – конец цепи, там свободная OH-группа
Представьте цепочку вагонов: 5′-конец – это локомотив, 3′-конец – последний вагон. Все ферменты “читают” ДНК в одном направлении (от 5′ к 3′), как поезд едет от головы к хвосту.
Две цепи ДНК идут “навстречу” друг другу:
5'-ATGCCTGA-3' → (прямая цепь)
||||||||
3'-TACGGACT-5' ← (комплементарная)
Когда мы говорим “3′-конец gRNA”, имеем в виду ту часть, которая ближе к PAM. Это важно для скоринга – скоро увидите почему.
Скоринг: не все gRNA одинаково полезны
В первой части мы отбирали gRNA по двум критериям: GC от 40 до 60% и отсутствие повторов. Но внутри этого диапазона кандидаты сильно различаются по эффективности.
Представьте, что вы выбираете сотрудника. Формально все кандидаты подходят (есть диплом, опыт работы), но кто-то явно лучше, поэтому нужна система оценки.
Что влияет на качество gRNA
GC-состав около 50% – золотая середина. Слишком много GC – цепи слипаются слишком крепко, gRNA не хочет отпускать ДНК после разрезания. Слишком мало – связывание слабое, Cas9 может “соскочить” раньше времени.
Нуклеотиды на концах – 3′-конец gRNA (ближе к PAM) критичен для связывания. Если там много G и C, комплекс получается слишком стабильным – как клей, который схватился намертво. Cas9 режет, но не может отцепиться и пойти дальше.
Первый нуклеотид – gRNA обычно экспрессируют с U6-промотора, а он капризный. Если первая буква – T (в РНК это U), промотор работает плохо. Если G – отлично.
Формула скоринга
Начинаем со 100 очков и вычитаем штрафы:
def calculate_score(sequence: str, gc_content: float, has_repeats: bool) -> float:
"""
Оценивает качество gRNA.
100 = идеал, штрафы уменьшают скор.
Бонус за G в начале может дать >100.
"""
score = 100.0
# Штраф за отклонение от оптимального GC (50%)
# Каждый процент отклонения = -1.5 очка
score -= abs(gc_content - 50) * 1.5
# повторы - это плохо
if has_repeats:
score -= 20
# Проверяем 3'-конец (последние 6 нуклеотидов)
last_6 = sequence[-6:]
gc_at_end = last_6.count('G') + last_6.count('C')
if gc_at_end > 3:
score -= (gc_at_end - 3) * 5
# Первый нуклеотид
if sequence[0] == 'T':
score -= 10 # U6 не любит тимин
elif sequence[0] == 'G':
score += 5 # U6 любит гуанин
return max(0, score)
Это упрощённая модель. Настоящие алгоритмы вроде Rule Set 2 используют машинное обучение на тысячах экспериментов, учитывают позицию каждого нуклеотида и ещё кучу факторов. Но для понимания принципа – достаточно.
Off-target: ищем случайных жертв
Самая большая проблема CRISPR специфичность. Геном человека – это три миллиарда пар нуклеотидов. Наша gRNA составляет всего 20 букв. Какова вероятность, что такая последовательность встретится где-то ещё?
Спойлер: высокая. И даже если точного совпадения нет, Cas9 может разрезать участки с 1-2 отличиями. Представьте, что вы ищете в толпе человека по описанию “высокий брюнет в синей куртке”. Найдёте нужного, но по дороге обратите внимание на пару похожих.
Для поиска таких “похожих” используют BLAST.
Что такое BLAST
BLAST (Basic Local Alignment Search Tool) – это Google для биологов. Вы даёте ему последовательность, он ищет похожие в гигантских базах данных: геномах, транскриптомах, протеомах.
Алгоритм хитрый: вместо того чтобы сравнивать каждую букву (это заняло бы вечность), BLAST сначала ищет короткие “слова” такие совпадения из 7-11 букв. Нашёл слово и расширяет в обе стороны, проверяя, насколько длинное совпадение получится.
В ответ отдаёт список отобранных и метрики:
-
E-value – вероятность случайного совпадения. E-value = 0.001 значит “такое совпадение случайно встретится раз на 1000 запросов”. Чем меньше – тем надёжнее.
-
Identity – процент идентичных букв. 100% = точное совпадение.
-
Coverage – какая часть запроса покрыта совпадением.
Biopython: швейцарский нож биоинформатика
Biopython – библиотека, которая умеет почти всё: читать форматы последовательностей, работать с базами NCBI, запускать BLAST, строить филогенетические деревья и многое другое.
Нам нужны два модуля:
-
Bio.Blast.NCBIWWW– отправляет запросы на сервер NCBI -
Bio.Blast.NCBIXML– парсит результаты
pip install biopython
Проверка off-target через BLAST
from Bio.Blast import NCBIWWW, NCBIXML
def check_off_targets(
sequence: str,
organism: str = "Homo sapiens",
max_hits: int = 50,
) -> list[dict]:
"""
Ищет потенциальные off-target через NCBI BLAST.
Внимание: запрос занимает 30-60 секунд!
NCBI просит не частить - максимум 1 запрос в 10 секунд.
"""
# Добавляем PAM к запросу
query = sequence + "NGG"
result_handle = NCBIWWW.qblast(
program="blastn", # нуклеотидный поиск
database="nt", # nucleotide collection
sequence=query,
entrez_query=f'"{organism}"[organism]',
hitlist_size=max_hits,
word_size=7, # для коротких последовательностей
expect=1000, # высокий порог, чтобы поймать слабые совпадения
megablast=False
)
off_targets = []
for record in NCBIXML.parse(result_handle):
for alignment in record.alignments:
for hsp in alignment.hsps:
identity = (hsp.identities / hsp.align_length) * 100
off_targets.append({
'title': alignment.title[:80],
'identity': round(identity, 1),
'mismatches': hsp.align_length - hsp.identities,
'e_value': hsp.expect,
})
result_handle.close()
return off_targets
Пара замечаний:
word_size=7 – обычно BLAST ищет “слова” из 11 букв, но наш запрос всего 23 буквы. С большим word_size он может ничего не найти.
expect=1000 – нарочно высокий порог. Для off-target нас интересуют даже слабые совпадения: участок с 85% идентичности всё ещё может быть разрезан Cas9.
Оценка риска
Что считать опасным?
-
≥95% идентичности – Cas9 скорее всего разрежет
-
85-95% – может разрезать, зависит от позиции мисматчей
-
<85% – обычно безопасно
def assess_off_target_risk(
off_targets: list[dict],
target_gene: str
) -> dict:
"""Сортирует off-target по уровню риска."""
# Паттерны для фильтрации целевого гена
short_name = target_gene.split()[0].split('_')[0].lower()
target_patterns = [
short_name,
"chemokine receptor 5",
"cc chemokine receptor",
"c-c motif chemokine receptor",
"ccr-5",
"nonfunctional cc chemokine",
"cc chemokine re",
]
high_risk = []
medium_risk = []
low_risk = []
for ot in off_targets:
title_lower = ot['title'].lower()
# Пропускаем сам целевой ген - это не off-target
if any(p in title_lower for p in target_patterns):
continue
if ot['identity'] >= 95:
high_risk.append(ot)
elif ot['identity'] >= 85:
medium_risk.append(ot)
else:
low_risk.append(ot)
return {
'high_risk': high_risk,
'medium_risk': medium_risk,
'low_risk': low_risk,
'is_safe': len(high_risk) == 0,
}
Да-да, знаю, что target_patterns убивает переиспользование для других генов, не CCR5, но для примера никакого простого способа собрать все синонимы, по имени из FASTA файла, я не нашёл :(
Собираем всё вместе
Код второй части живёт в отдельном файле и импортирует функции из первой:
# crispr_finder_v2.py
from pathlib import Path
from Bio.Blast import NCBIWWW, NCBIXML
import typer
from crispr_finder import (
parse_fasta,
analyze_sequence,
filter_candidates,
)
app = typer.Typer()
def calculate_score(sequence: str, gc_content: float, has_repeats: bool) -> float:
"""Оценивает качество gRNA по шкале 0-100+."""
score = 100.0
score -= abs(gc_content - 50) * 1.5
if has_repeats:
score -= 20
last_6 = sequence[-6:]
gc_at_end = last_6.count('G') + last_6.count('C')
if gc_at_end > 3:
score -= (gc_at_end - 3) * 5
if sequence[0] == 'T':
score -= 10
elif sequence[0] == 'G':
score += 5
return max(0, score)
def score_candidates(candidates: list[dict]) -> list[dict]:
"""Добавляет скор к каждому кандидату."""
for c in candidates:
c['score'] = calculate_score(
c['sequence'],
c['gc_content'],
c['has_poly_repeats']
)
return candidates
def check_off_targets(
sequence: str,
organism: str = "Homo sapiens",
max_hits: int = 50,
) -> list[dict]:
"""Ищет off-target через NCBI BLAST."""
query = sequence + "NGG"
result_handle = NCBIWWW.qblast(
program="blastn",
database="nt",
sequence=query,
entrez_query=f'"{organism}"[organism]',
hitlist_size=max_hits,
word_size=7,
expect=1000,
megablast=False
)
off_targets = []
for record in NCBIXML.parse(result_handle):
for alignment in record.alignments:
for hsp in alignment.hsps:
identity = (hsp.identities / hsp.align_length) * 100
off_targets.append({
'title': alignment.title[:80],
'identity': round(identity, 1),
'mismatches': hsp.align_length - hsp.identities,
'e_value': hsp.expect,
})
result_handle.close()
return off_targets
def assess_off_target_risk(off_targets: list[dict], target_gene: str) -> dict:
"""Оценивает риск off-target эффектов."""
short_name = target_gene.split()[0].split('_')[0].lower()
target_patterns = [
short_name,
"chemokine receptor 5",
"c-c motif chemokine receptor 5",
]
high_risk = []
medium_risk = []
low_risk = []
for ot in off_targets:
title_lower = ot['title'].lower()
if any(p in title_lower for p in target_patterns):
continue
if ot['identity'] >= 95:
high_risk.append(ot)
elif ot['identity'] >= 85:
medium_risk.append(ot)
else:
low_risk.append(ot)
return {
'high_risk': high_risk,
'medium_risk': medium_risk,
'low_risk': low_risk,
'is_safe': len(high_risk) == 0,
}
@app.command()
def analyze(
fasta_path: Path = typer.Argument(..., help="Путь к FASTA файлу"),
gc_min: float = typer.Option(40.0, help="Минимальный GC-состав"),
gc_max: float = typer.Option(60.0, help="Максимальный GC-состав"),
top_n: int = typer.Option(10, help="Показать топ N кандидатов"),
check_offtarget: bool = typer.Option(False, help="Проверить off-target (медленно!)"),
):
"""Анализ gRNA с скорингом и опциональной проверкой off-target."""
sequences = parse_fasta(fasta_path)
for name, sequence in sequences.items():
typer.echo(f"n{'=' * 60}")
typer.echo(f"Ген: {name}")
typer.echo(f"Длина: {len(sequence)} нуклеотидов")
typer.echo(f"{'=' * 60}")
# Ищем и фильтруем кандидатов (функции из первой части)
candidates = analyze_sequence(sequence)
filtered = filter_candidates(candidates, gc_min, gc_max, allow_repeats=False)
# Добавляем скоринг
scored = score_candidates(filtered)
scored.sort(key=lambda x: x['score'], reverse=True)
if not scored:
typer.echo("Кандидатов не найдено")
continue
typer.echo(f"nНайдено кандидатов: {len(scored)}")
typer.echo(f"Топ-{min(top_n, len(scored))} по скору:n")
for i, c in enumerate(scored[:top_n], 1):
strand = "+" if c['is_strand'] else "-"
typer.echo(
f"{i:2}. {c['sequence']} | "
f"score: {c['score']:.1f} | "
f"GC: {c['gc_content']:.1f}% | "
f"strand: {strand}"
)
if check_offtarget:
typer.echo(" Проверяю off-target...")
try:
off_targets = check_off_targets(c['sequence'])
risk = assess_off_target_risk(off_targets, name)
if risk['is_safe']:
typer.echo(" ✓ Высокий риск off-target не обнаружен")
else:
typer.echo(
f" ⚠ Риск: {len(risk['high_risk'])} высокий, "
f"{len(risk['medium_risk'])} средний"
)
for ot in risk['high_risk'][:3]:
typer.echo(f" - {ot['title']}: {ot['identity']}%")
except Exception as e:
typer.echo(f" ✗ Ошибка: {e}")
if __name__ == "__main__":
app()
Тестируем на CCR5
Скачиваем последовательность CCR5 (или берём из репозитория) и запускаем:
python crispr_finder_v2.py analyze ccr5.fasta --top-n 5
============================================================
Ген: CCR5_CDS Homo sapiens C-C motif chemokine receptor 5 (CCR5), mRNA
Длина: 1059 нуклеотидов
============================================================
Найдено кандидатов: 62
Топ-5 по скору:
1. GTCATGGTCATCTGCTACTC | score: 105.0 | GC: 50.0% | strand: +
2. GACAAGTGTGATCACTTGGG | score: 100.0 | GC: 50.0% | strand: +
3. ATGCAGGTGACAGAGACTCT | score: 100.0 | GC: 50.0% | strand: +
4. CAGTTTACACCCGATCCACT | score: 100.0 | GC: 50.0% | strand: +
5. GTAAACTGAGCTTGCTCGCT | score: 100.0 | GC: 50.0% | strand: -
Первый кандидат набрал 105 очков – больше “максимума”. Это бонус за гуанин в начале: такие gRNA отлично экспрессируются с U6-промотора.
С флагом --check-offtarget программа проверит каждого кандидата через BLAST. Это займёт несколько минут (NCBI не торопится), но покажет, есть ли в геноме человека опасные похожие участки.
python crispr_finder_v2.py analyze ccr5.fasta --top-n 5 --check-offtarget
============================================================
Ген: CCR5_CDS Homo sapiens C-C motif chemokine receptor 5 (CCR5), mRNA
Длина: 1059 нуклеотидов
============================================================
Найдено кандидатов: 62
Топ-5 по скору:
1. GTCATGGTCATCTGCTACTC | score: 105.0 | GC: 50.0% | strand: +
Проверяю off-target (займёт ~30-60 сек)...
✓ Высокий риск off-target не обнаружен
2. GACAAGTGTGATCACTTGGG | score: 100.0 | GC: 50.0% | strand: +
Проверяю off-target (займёт ~30-60 сек)...
✓ Высокий риск off-target не обнаружен
3. ATGCAGGTGACAGAGACTCT | score: 100.0 | GC: 50.0% | strand: +
Проверяю off-target (займёт ~30-60 сек)...
✓ Высокий риск off-target не обнаружен
4. CAGTTTACACCCGATCCACT | score: 100.0 | GC: 50.0% | strand: +
Проверяю off-target (займёт ~30-60 сек)...
✓ Высокий риск off-target не обнаружен
5. GTAAACTGAGCTTGCTCGCT | score: 100.0 | GC: 50.0% | strand: -
Проверяю off-target (займёт ~30-60 сек)...
✓ Высокий риск off-target не обнаружен
Что можно запилить далее
Мы написали рабочий инструмент, но до уровня Benchling или CRISPOR ему далеко:
-
ML-скоринг – использовать модели типа Azimuth, обученные на реальных экспериментах
-
Локальный BLAST – скачать геном и искать без интернета и лимитов NCBI
-
Позиция мисматчей – мисматч рядом с PAM опаснее, чем на 5′-конце
-
Визуализация – карта гена с отмеченными сайтами
-
Batch-режим – много генов за раз с кэшированием
Но для понимания принципов – достаточно.
Итого
За две статьи мы:
-
Разобрались, как работает CRISPR-Cas9
-
Написали поиск PAM и извлечение gRNA
-
Добавили фильтрацию и скоринг
-
Подключили BLAST для проверки off-target
-
Протестировали на настоящем гене CCR5
Код в репозитории. Там же FASTA с CCR5.
Послесловие
Как я писал в комментариях к первой части, есть домашние лабораторные наборы с помощью которых можно сделать, например, светящееся пиво или что-то тревожно напоминающее биологическое оружие бактерию выживающую в стрептомицине (антибиотике). Если интересно про что-то такое (и сбоку пайтон) почитать, то голосуйте в опросе под статьёй, устрою в ближайшем будущем (постараюсь).
P.S. Биологам: простите за упрощения. Цель – показать принцип, а не заменить специализированные инструменты.
P.P.S. Не редактируйте людей. Даже если очень хочется. Особенно если очень хочется.
P.P.P.S. Обсудить или покритиковать – велком в телеграм.
Автор: egnodus


